14+NC,全代码,完整复现!实用!(2)
【作者】:网站采编
【关键词】:
【摘要】最后使用run_lm_stats_limma()函数拟合线性模型。 结果显示: 在5.2中重现图5G? 首先,我们从线索.io数据库下载药物和工具化合物注释,然后选择药物名称、
最后使用run_lm_stats_limma()函数拟合线性模型。
结果显示:
在5.2中重现图5G?
首先,我们从线索.io数据库下载药物和工具化合物注释,然后选择药物名称、靶点等相关信息。
结果显示:
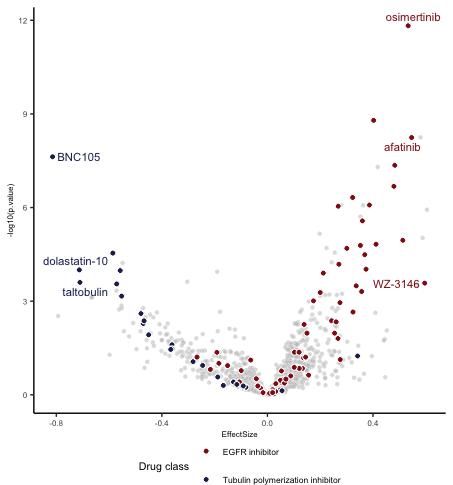
根据药物相关的注释信息,将线性拟合结果lmstats_out_chemical_prism与注释结果相结合,筛选出EGFR或Tubulin靶向的药物。
准备好输入数据后,使用ggplot()函数绘制火山图并添加标签,就像图5E的绘制一样。
结果显示:
 六、图5H的再现
六、图5H的再现
在正式绘制之前,很自然的清理和准备输入数据。
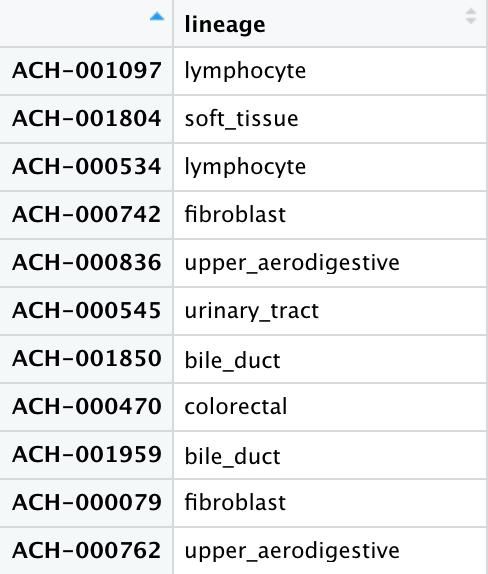
首先选择细胞系的源组织及其对应的细胞名,生成新的数据文件covars.
结果显示:
 同时使用intersect()函数得到第12个CRISPR的共同细胞系名称common_CLs和第28簇中的注释信息比对和敲除数据。结果显示,最终得到了684个共同细胞系。
同时使用intersect()函数得到第12个CRISPR的共同细胞系名称common_CLs和第28簇中的注释信息比对和敲除数据。结果显示,最终得到了684个共同细胞系。
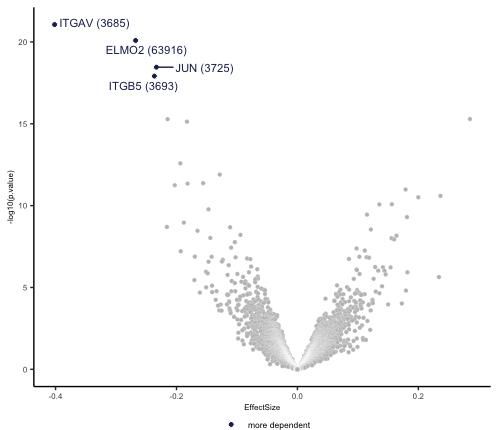
最后使用run_lm_stats_limma()函数对其进行线性拟合,得到图5H的输入数据。
6.2?复发图5H
同时,相关药物的标签设置。
结果显示:
结果显示:
 这里,整篇Nature Communication转载~整篇文章基于肿瘤样本和细胞系的表达数据,使用Cellinger进行数据分析,使用ggplot()函数进行可视化展示。
这里,整篇Nature Communication转载~整篇文章基于肿瘤样本和细胞系的表达数据,使用Cellinger进行数据分析,使用ggplot()函数进行可视化展示。
在首页回复“阿琛66”获取本次数据和代码~
系列门户
—END—
< p >作者丨A?陈排版丨司锦兄弟
主编丨雪秋
文章来源:《中国实用医药》 网址: http://www.zgsyyyzzs.cn/zonghexinwen/2021/0724/1615.html
中国实用医药投稿 | 中国实用医药编辑部| 中国实用医药版面费 | 中国实用医药论文发表 | 中国实用医药最新目录
Copyright &copy; 20019-2020 中国实用医药 版权所有
投稿电话: 投稿邮箱:

